BSTViewer_v3.0软件安装图文教程

BSTMatrix v2.3.R环境搭建、安装及使用说明
2022年6月8日
新品上线 | 百创DG1000单细胞转录组测序重磅来袭
2022年6月20日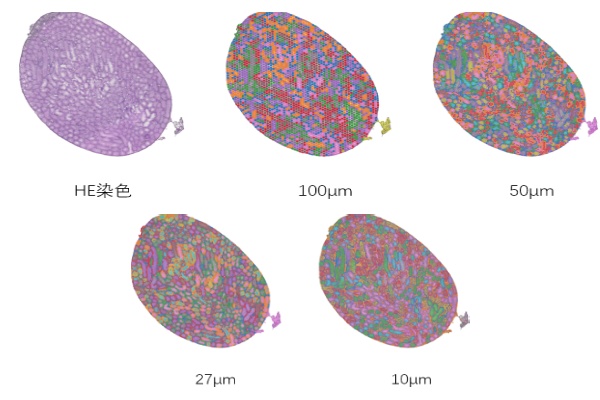
操作步骤
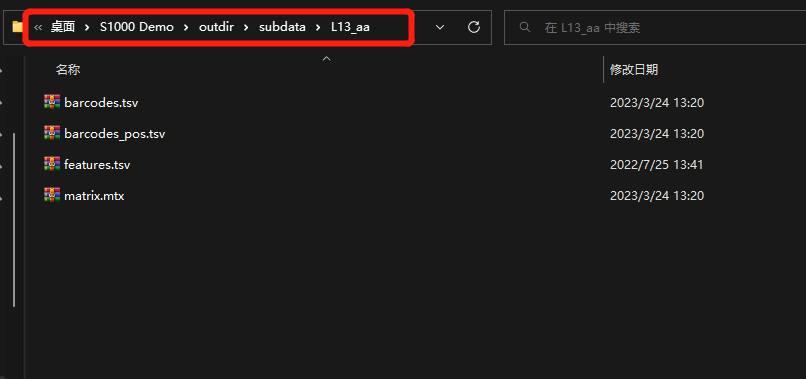
1)将官网下载到的BSTViewer_project文件夹数据(或者通过运行BSTMatrix 得到的BSTViewer_project 文件夹)存放到英文命名的文件夹之中;【包括各个级别分辨率矩阵文件,及BMChiper 或者官网下载的HE 染色图片】可以同时在此文件夹下边新建一个outdir 作为文件输出路径;
2)运行“BSTViewer V3.0正式版-WIN-64\BSTViewer V3.0正式版\bin”目录下的BSTViewer,点击“创建项目”创建任务,选择1)中的文件夹outdir;
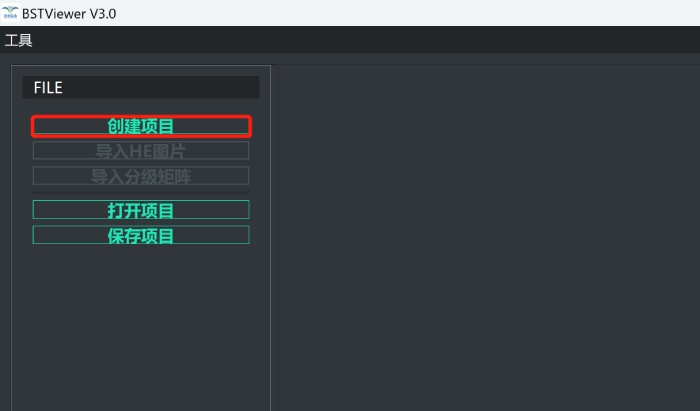
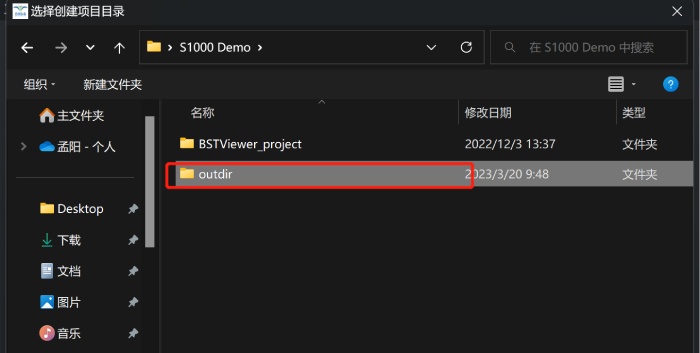
创建输出目录
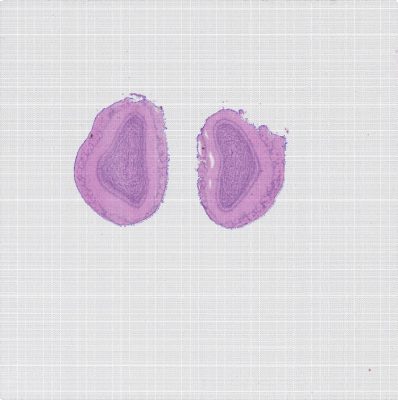
3)导入HE图片和矩阵文件。导入的HE图片可以通过鼠标左键进行移动,滚轮可对图片进行缩放;
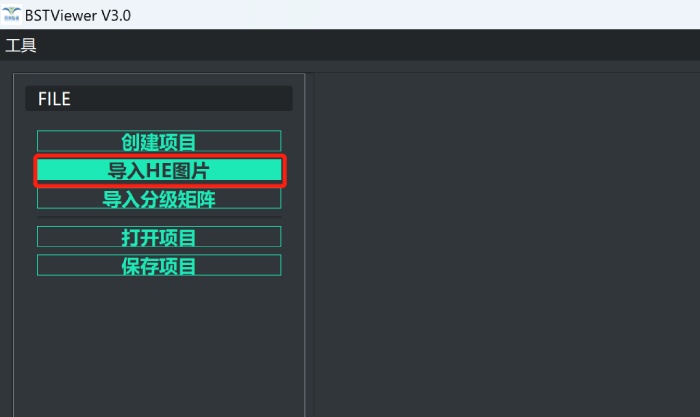
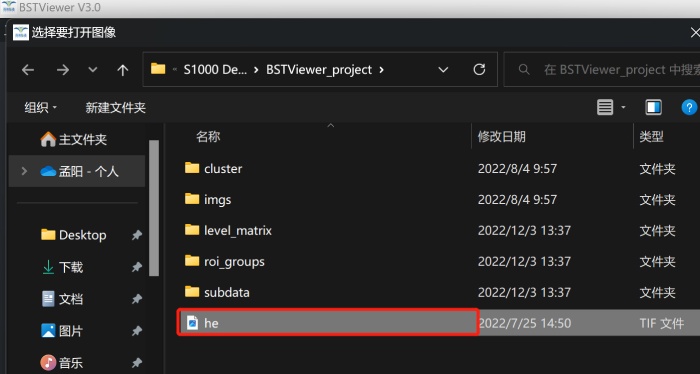
导入HE图片
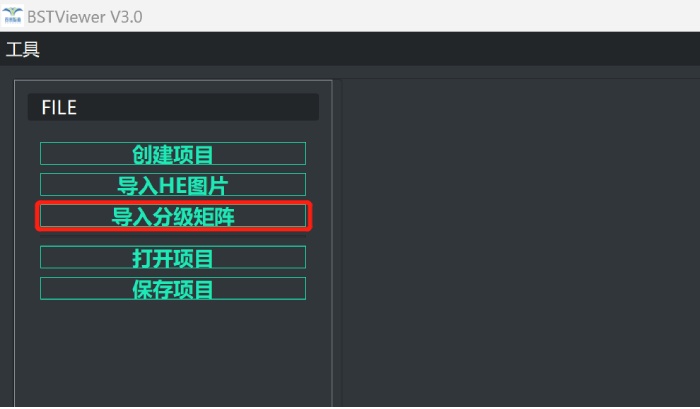
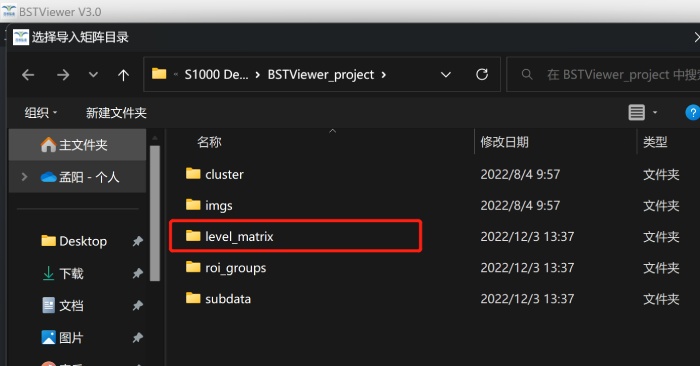
导入矩阵
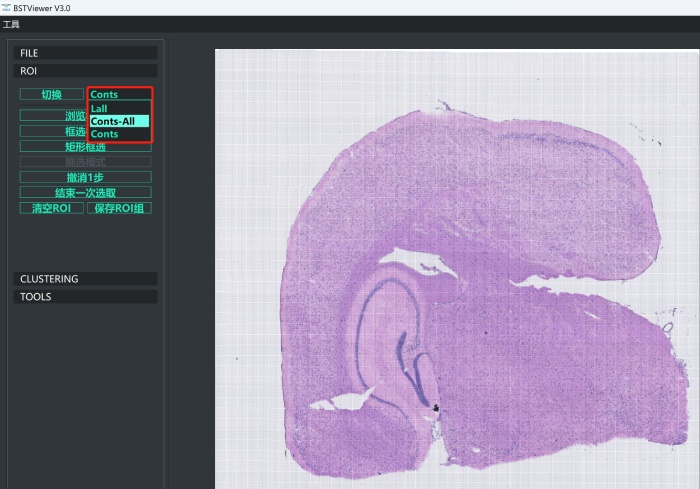
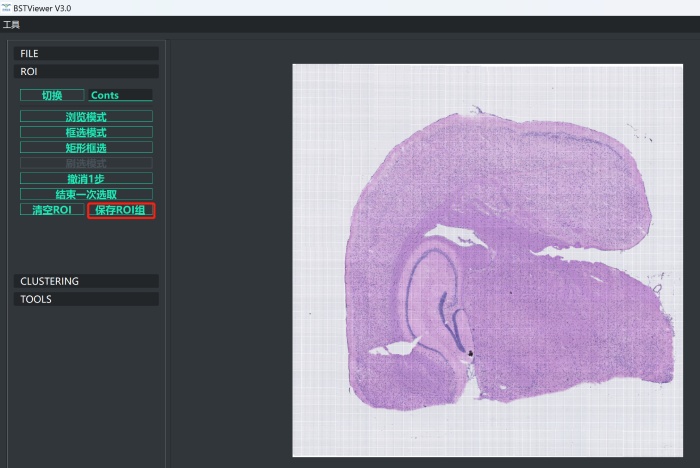
4)进行ROI 创建。点击“ROI”按钮,此处可以选择全局聚类和部分区域聚类。cont和conts-all分别代表在全局下聚类与全局扣除空白区域聚类。区域聚类的话目前提供了“框选模式”和“矩形框选”两种模式。框选合适的区域后点击“结束一次选取”,选择“保存或抠除”,完成之后点击“保存ROI组”并命名即可;

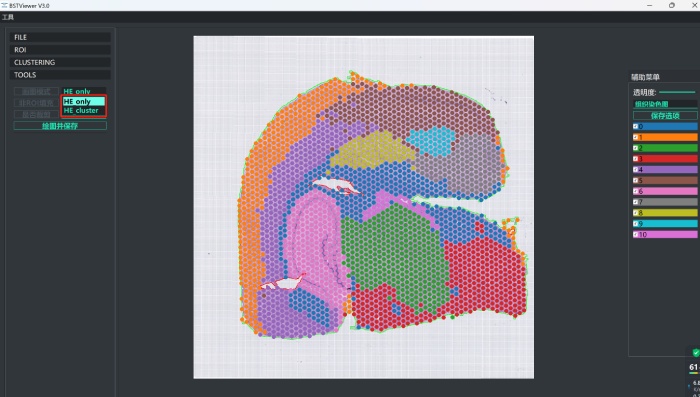
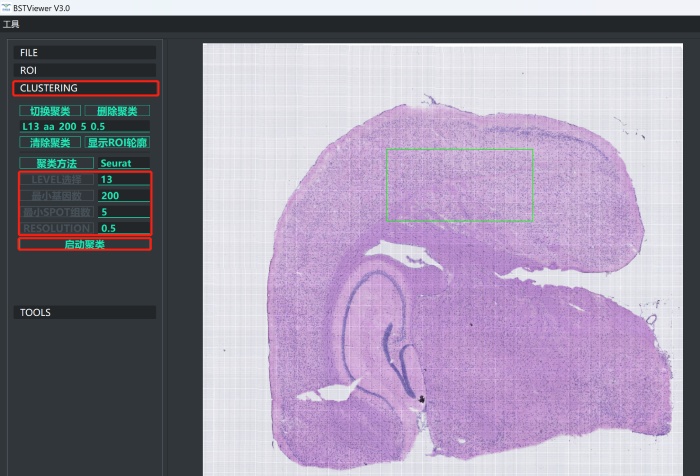
5)点击“Clustering”,选取自己感兴趣的分辨率,和适当的参数,然后点击“启动聚类”;
LEVEL选择:不同等级分辨率
最小基因数:当单个spot中基因数少于这个数目则会被自动过滤掉;
最小spot组数:单个基因需要表达的spot数,少于这个数目该基因会被过滤掉;
RESOLUTION:聚类丰富度值,该数值越高cluster种类越多,推荐在0.5附近调整。
区域框选
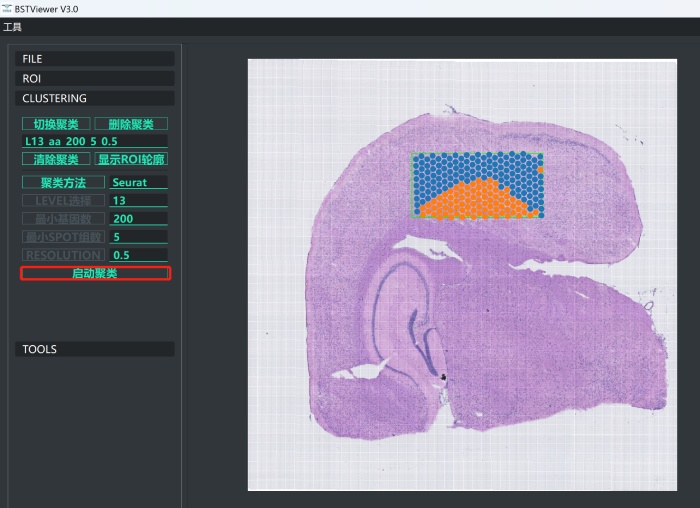
区域聚类结果

全局聚类结果
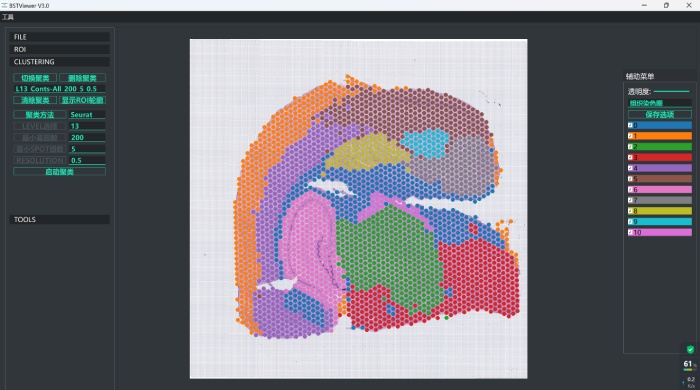
抠除空白的全局聚类结果
6)最后得到框选到的感兴趣区域的聚类结果,点击“ROI”切换为“浏览模式”可以通
过鼠标滚轮进行缩放,查看结果;
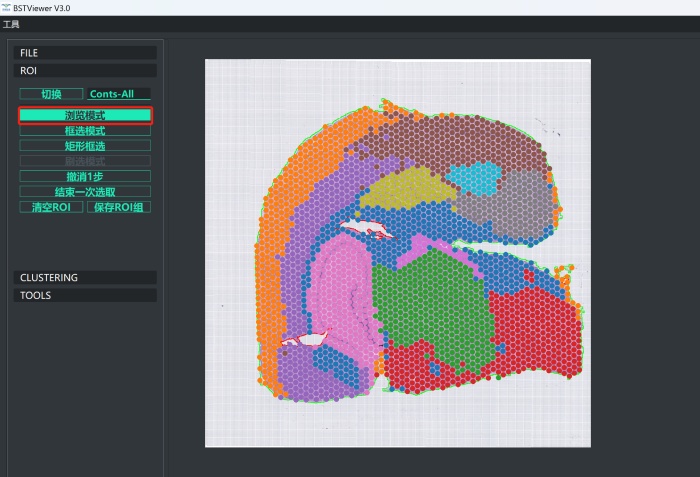
7)聚类图片可以在TOOLS栏中进行保存。点击图画模式可以选择“仅保存HE”和“HE&Cluster”分别进行保存。
结果路径
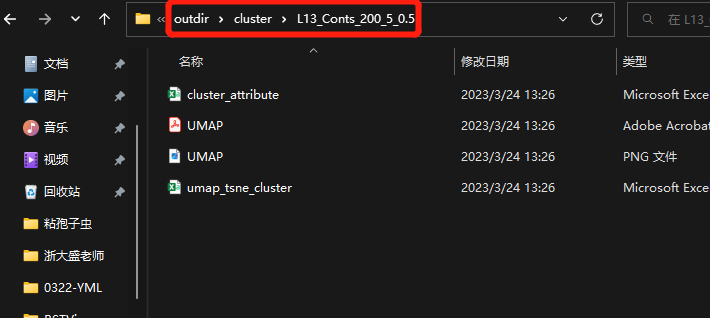
1)cluster聚类结果保存在“outdir文件夹”中的“cluster”中。包含cluster聚类图片、barcode信息、UMAP、tSNE聚类结果。
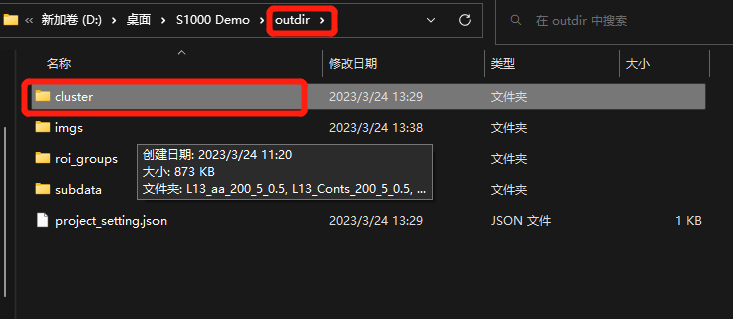
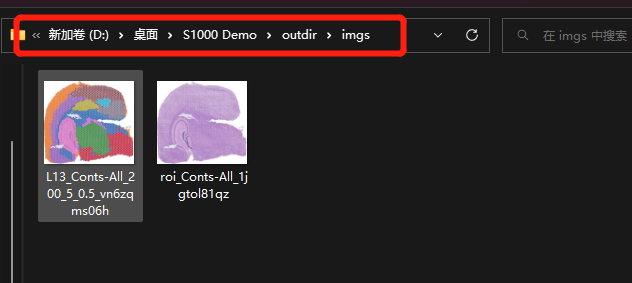
2)聚类过程中保存的HE图片和cluster聚类图片保存在“imgs”文件夹中。
3)选择的区域聚类的矩阵文件保存在“subdata”文件夹中