BSTMatrixONT流程说明

Bmk_Space_mapping.R 操作说明
2022年8月25日
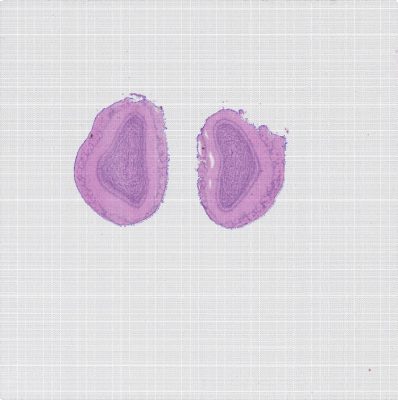
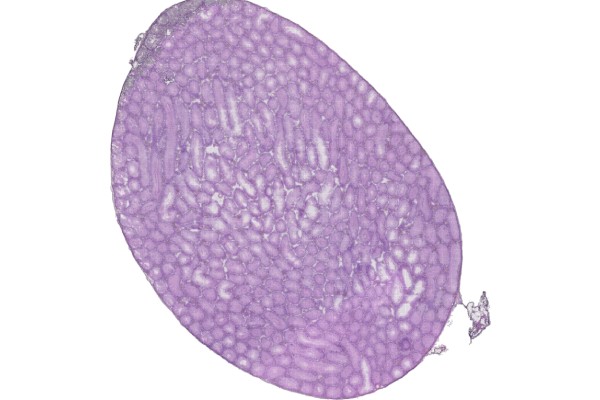
BMKMANU S1000小鼠胚胎头部Demo数据结果展示
2022年11月23日
1. 软件依赖及安装
依赖:
- python: 版本9,安装cv2模块(4.0以上版本),Cython,numpy,seaborn模块
- Minimap2
- Samtools:版本2及以上
依赖的软件需要使用export添加到环境变量中,以实现流程的调用。
安装:在流程目录下运行以下命令
pip install ./bmk-ocean-stars
2. 输入数据准备
- 测序数据:ONT测序fastq数据(支持.gz格式)。
- 参考基因组数据:基因组fa序列文件,gtf文件(包含gene、exon)。
- 荧光解码文件及HE图片文件。
3. 配置文件编写
配置文件:
## 测序数据
FQ /path/to/ont-reads.fastq
## 荧光解码文件
FLU /path/to/flu_info.txt
## 参考基因组序列、gtf文件
Genome /path/to/ref/genome/fasta
GTF /path/to/ref/gene/gtf
## HE图片文件
HE /path/to/HE.tif
## 输出目录及输出前缀
OUTDIR /path/to/result/dir/
PREFIX outfile-prefix
### 程序运行线程数
Threads 8
Link1 TACGT
4. 流程运行
- 流程说明:
流程分为4个步骤,如下所示:
- 步骤1:运行GenomeMap,将ONT reads与参考基因组比对。
- 步骤2:运行MatrixMake,识别barcode、UMI并生成表达矩阵文件。
- 步骤3:运行AllheStat,处理HE图片。
- 步骤4:运行WebReport,得到网页版报告。
- 流程参数:
-c config.txt 数据配置文件
-s 步骤选择,0为运行1-4所有步骤,也可选择个别步骤单独运行,多个步骤中间使用“,”分割。
- 参考命令:
./BSTMatrixONT -c config.txt -s 0
./BSTMatrixONT -c config.txt -s 1,2,3,4
./BSTMatrixONT -c config.txt -s 1,2
5. 结果文件说明
- 目录结构及结果说明:
outdir/
├── 01.GenomeMap 步骤1运行结果目录
│ ├── HXFA2-3.sort.bam 基因组比对bam文件
│ ├── HXFA2-3.sort.bam.bai
│ └── mapping.sh
├── 02.MatrixMake 步骤2运行结果目录
│ ├── bam
│ ├── bc_umi_read.tsv barcode-umi-reads数文件
│ ├── bc_umi_read.tsv.stat
│ ├── cache_raw_expression.pkl
│ ├── filtered_feature_bc_matrix
│ ├── final_matrix 过滤后的matrix文件
│ ├── flu_recognition barcode芯片位置识别目录
│ ├── HXFA2-3.bc_gene_umi.bc_stat barcode检测结果统计文件
│ ├── HXFA2-3.bc_gene_umi.map_stat 比对类型结果统计文件
│ ├── HXFA2-3.bc_gene_umi.stat barcode-gene-umi对应文件
│ ├── raw_feature_bc_matrix
│ ├── reads_cumi.tsv barcode-gene-umi数文件
│ ├── reads_info.tsv reads相关信息文件
│ └── web_summary.html 网页版统计结果文件
├── 03.AllheStat 步骤3运行结果目录
│ ├── allhe
│ ├── all_level_stat.txt 不同水平的spots统计
│ ├── BSTViewer_project BSTViewer软件输入数据
│ ├── heAuto_level_matrix
│ ├── level_matrix
│ ├── stat.txt
│ └── umi_plot umi count图片统计图目录
├── 04.WebReport 步骤4运行结果目录
│ ├── prefix.filelist
│ ├── index.html 网页版报告html文件
│ └── src 网页版报告src目录
└── prefix 收集的基因表达矩阵等文件目录
├── barcode_pos.tsv barcode类型对应的芯片位置文件
├── barcode.tsv 芯片对应的barcode类型文件
├── bc_gene_umi.stat.gz barcode-gene-umi对应文件
├── bc_umi_read.tsv.gz barcode-umi-reads数文件
├── features.tsv features.tsv文件
└── matrix.tsv 基因表达矩阵文件